迷你課程:R語言-9~程式效能測量與優化

Quick look
在 R 語言中,提高程式執行效率和減少記憶體使用是許多開發者關心的課題。透過測量程式的資源消耗,我們可以識別瓶頸,並集中精力進行優化。本教學將探討如何透過計時與性能分析工具(如 proc.time() 和 profvis)來測試程式效能,並透過不同的優化策略提升程式的執行速度。
R 語言效能優化的基本概念
向量化操作比迴圈快
避免動態增長物件
使用適當的數據結構
利用內建函數而非 apply()
有效利用
data.table和 dplyr使用
profvis來找出效能瓶頸
測量程式執行時間
x <- rnorm(1e7)
# 使用 for 迴圈計算 exp(x)
t1 <- proc.time()
y <- rep(NA, length(x))
for (j in 1:length(x)) {
y[j] <- exp(x[j])
}
t2 <- proc.time()
# 使用向量化操作計算 exp(x)
y <- exp(x)
t3 <- proc.time()
# 印出執行時間
print(t2 - t1) # for 迴圈方式
print(t3 - t2) # 向量化方式
結果顯示向量化操作大幅提升執行效率。
避免動態增長物件
x <- rnorm(1e5)
t1 <- proc.time()
y <- c()
for (j in 1:length(x)) {
y <- c(y, exp(x[j]))
}
t2 <- proc.time()
print(t2 - t1)
這種方式會頻繁重新分配記憶體,應改用預先分配 NA 的向量。
x <- rnorm(1e5)
y <- numeric(length(x))
t1 <- proc.time()
y <- exp(x)
t2 <- proc.time()
print(t2 - t1)
使用適當的數據結構
n <- 8000
X <- matrix(rnorm(n*n), n, n)
D <- as.data.frame(X)
t1 <- proc.time()
for (j in 1:10000) {
i1 <- sample(1:n, 1)
i2 <- sample(1:n, 1)
X[i1, i2] <- exp(X[i1, i2])
}
t2 <- proc.time()
print(t2 - t1)
t1 <- proc.time()
for (j in 1:10000) {
i1 <- sample(1:n, 1)
i2 <- sample(1:n, 1)
D[i1, i2] <- exp(D[i1, i2])
}
t2 <- proc.time()
print(t2 - t1)
結果顯示,矩陣 (matrix) 的操作比 data.frame 快許多。
使用內建函數而非 apply()
t1 <- proc.time()
y <- apply(X, 2, sum)
t2 <- proc.time()
y <- colSums(X)
t3 <- proc.time()
print(t2 - t1)
print(t3 - t2)
內建函數 colSums() 比 apply() 更快。
使用profvis分析效能
profvis 是 R 的效能分析工具,可以幫助我們找出程式的效能瓶頸。以下提供一個進階的示例,涵蓋 資料處理 和 模型訓練,並透過 profvis 來識別可能的效能問題。
情境:大規模資料處理與回歸分析
我們將使用 mtcars 資料集(擴展為 10 萬筆資料),進行資料前處理、特徵工程,並使用 lm() 進行線性回歸。
library(profvis)
library(dplyr)
# 創建較大的數據集
set.seed(123)
big_data <- mtcars[rep(1:nrow(mtcars), times = 5000), ] # 擴展為 10 萬筆資料
big_data$id <- 1:nrow(big_data) # 添加唯一 ID
big_data$category <- sample(letters[1:5], nrow(big_data), replace = TRUE) # 添加分類變數
# 使用 profvis 來分析效能瓶頸
profvis({
# 1. 數據處理:篩選、高效轉換、合併
filtered_data <- big_data %>%
filter(mpg > 15 & hp < 200) %>%
mutate(log_disp = log(disp + 1), # 避免 log(0)
category = as.factor(category)) %>%
arrange(desc(mpg)) # 按 mpg 排序
# 2. 建立新特徵
grouped_data <- filtered_data %>%
group_by(category) %>%
summarise(mean_mpg = mean(mpg), mean_hp = mean(hp), .groups = "drop")
# 3. 進行線性回歸分析
model <- lm(mpg ~ hp + wt + log_disp, data = filtered_data)
summary(model)
# 4. 預測(使用模型進行預測)
predictions <- predict(model, newdata = filtered_data)
})
效能分析與優化建議
觀察 profvis 報告
profvis() 會顯示不同函數的 CPU 和記憶體使用情況。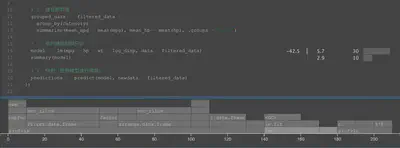
解釋
lm() 佔據了一定的計算時間,並且 model.frame.default 也花費了 20 ms,表示模型構建時數據準備(轉換 data frame)較為耗時。 summary() 花費 10 ms,但這部分通常不會影響核心計算。
優化建議:
考慮改用 lm.fit()
- lm() 內部會先建立 model.frame(),這個過程較為緩慢。如果數據已經是合適格式,使用 lm.fit() 可以略微加速計算。
減少 data.frame 轉換時間如果 model.frame.default 佔據較多時間,可以確保變數格式是數值型 (numeric) 或因子 (factor),而非 character,這樣 lm() 執行時能夠更高效處理數據。- filter() 和 arrange() 可能是耗時的操作,建議優先使用 data.table 來加速篩選與排序。
- 使用
data.table加速資料處理 - 使用 fread() 讀取大檔案
- 避免不必要的 factor 轉換
- 嘗試 lm.fit() 來取代 lm() 以加速線性回歸
我們來試者改改看將lm換成用lm.fit()會不會提升效能
profvis({
# 1. 數據處理:篩選、高效轉換、合併
filtered_data <- big_data %>%
filter(mpg > 15 & hp < 200) %>%
mutate(log_disp = log(disp + 1), # 避免 log(0)
category = as.factor(category)) %>%
arrange(desc(mpg)) # 按 mpg 排序
# 2. 建立新特徵
grouped_data <- filtered_data %>%
group_by(category) %>%
summarise(mean_mpg = mean(mpg), mean_hp = mean(hp), .groups = "drop")
# 3. 進行線性回歸分析(使用 lm.fit())
y <- filtered_data$mpg # 目標變數
X <- model.matrix(~ hp + wt + log_disp, data = filtered_data) # 創建設計矩陣
model <- lm.fit(X, y) # 直接傳入矩陣
coefficients <- model$coefficients # 取得迴歸係數
# 4. 預測(使用矩陣運算)
predictions <- X %*% coefficients
})
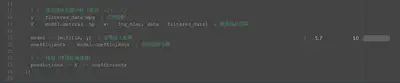
結果發現果然效率大幅提升!
課程小結
- 測量程式效能: 使用 proc.time() 和 profvis 來找出效能瓶頸。
- 向量化操作: 盡量避免使用 for 迴圈,優先考慮內建函數。
- 避免動態擴展向量: 先分配記憶體,再填充數據。
- 選擇合適的數據結構: matrix 比 data.frame 快。